ABSTRACTObjective:The etiology of the Kawasaki disease (KD) remains unknown despite of extensive studies but infection, immunity, and genetics were suggested as causes. There have been attempted to link susceptibility to KD to allelic variations to search related gene. The enabled homolog (Enah) gene on the human chromosome 1q42.12 encodes enabled/vasodilator-stimulated phosphoprotein (Ena/VASP). Ena/VASP is a regulator of actin cytoskeleton, exists in cytoplasm, and maintains homeostasis such as immune response, blood vessel preservation, and hemostasis. The aim of this study was to investigate polymorphisms of the Enah gene as a risk factor for KD and coronary artery lesions (CALs) as a complication.
Methods:In the Enah gene region, 15 single nucleotide polymorphisms (SNPs) were selected using human SNP websites (http://www.hapmap.org/, genome build). Three hundred and six healthy controls and 106 KD subjects were recruited. SNP genotyping was performed using the Golden Gate assay on an Illumina BeadStation 500 GX (Illumina Inc., San Diego, CA, USA). Frequencies of allele were obtained and the genetic association between of the Enah gene polymorphisms and susceptibility to KD and CALs was analyzed by SNPstats, Haploview software ver. 4.1 (Broad Institute, Cambridge, MA, USA). Multiple logistic regression analysis with adjustment for gender was performed.
서 론가와사키병은 전신을 침범하는 급성 열성 혈관염으로 소아에게서 발생하는 후천성 심질환의 가장 흔한 원인질환으로 알려져 있다. 관상동맥 병변이 발생할 수 있어 협심증, 심근경색, 급사 등 치명적인 상황이 발생하기도 한다[1]. 현재까지 광범위한 연구에도 불구하고 발병 원인은 정확히 알려져 있지는 않으나 감염, 면역, 유전관련설 등이 제시되고 있으며[2], 가와사키병의 발병이나 관상동맥류 발생에 취약한 유전자 다형성(gene polymorphism)에 대한 여러 후보 유전자들이 연구되어왔다[3].
Enabled homolog (Enah)는 enabled/vasodilator-stimulated phosphoprotein (Ena/VASP)을 암호화하는 유전자이다. Ena/VASP는 세포질에 존재하는 세포뼈대(cytoskeleton)의 구성성분인 액틴 세포뼈대의 조절자로 면역반응, 혈관의 유지, 지혈 등과 같은 인체 항상성을 유지하는 역할을 한다[4]. 저자는 감염 등 외부요인이 촉발하였을 때 Ena/VASP의 액틴 세포뼈대 조절의 잘못으로 면역매개성 염증으로 인한 혈관염이 생겨 가와사키병이 발생하고, 아울러 혈관벽 세포의 이동과 부착, 성장이 달라지고 혈관의 파괴와 변화, 섬유화 등으로 관상동맥 병변이 발생할 수 있다고 추정하였다. 이러한 가설하에 한국 아동에서 Enah 유전자 다형성과 가와사키병 발병, 관상동맥 병변 발생의 연관성 유무에 대해 알아보고자 본 연구를 시행하였다.
대상 및 방법1. 연구대상경희대학교병원 소아청소년과에 2003년 7월부터 2004년 12월까지 가와사키병으로 입원한 환아를 대상으로 하였다. 환아는 모두 106명(남아 72명, 여아 34명; 평균 연령 2년 8개월±2세)으로 진단기준으로는 5일 이상 지속되는 열과 다음의 5가지 동반증상 중 4항목 이상이 있으면 진단하였다. 첫째, 화농이 없는 양측 결막 충혈, 둘째, 입술의 홍조 및 균열, 딸기혀, 구강 발적, 셋째, 부정형 발진, 넷째, 급성기의 1.5 cm 이상 크기의 비화농성 경부 림프절 비대, 다섯째, 급성기 손발의 경성 부종과 홍조, 아급성기의 손톱, 발톱 끝부터 시작되는 막양 낙설이다. 단 관상동맥의 병변이 동반된 경우에는 3가지 조건만 충족하면 진단하였다[5].
치료로는 체중당 2 g의 면역글로불린을 1회 정주하였다. 정주 후 36시간 경과 후에도 열이 계속되면 면역글로불린을 재차 투여하였다.
관상동맥 합병을 보기 위해 면역글로불린 투여 시작 후 1-2일 내와 발병 시점에서 8주 후 심장초음파검사를 시행하였다. 관상동맥 병변은 일본 후생성 기준에 따라 5세 미만의 환아에서는 내경이 3mm를 초과하는 경우로 정의하였고, 5세 이상의 환아에서는 4mm를 초과하는 경우로 정의하였다. 또한 인접 부위 관상동맥 내경에 비해 1.5배 이상이거나 관상동맥 내강이 명백하게 불규칙한 경우도 관상동맥 병변으로 정의하였다[1].
정상 대조군은 건강한 지원자 306명(남자 134명, 여자 172명; 평균 연령 41±13세)을 대상으로 하였으며, 가와사키병, 자가면역질환, 항염증치료 중인 자, 고혈압, 고지질혈증, 뇌졸중, 동맥경화, 기타심장질환, 암, 혈전증, 신염 등의 병력이 있는 자는 제외하였다.
2. 연구방법유전자 DNA는 genomic DNA isolation reagent kit (CoreBio-System, Seoul, Korea)를 사용하여 말초혈액에서 얻었다. Enah 유전자 다형성이 가와사키병과 연관되어 있는지 알아보기 위해 선택된 단일염기다형성(single nucleotide polymorphisms, SNPs)으로 유전자형(genotype)을 결정하는 방법으로 연구하였다. SNPs는 사람 SNP 웹사이트(http://www.hapmap.org/, genome build)를 이용하여 Enah 유전자 부위에서 15개를 선택하였다. 이형접합(heterozygosity)이 알려져 있지 않거나 대립유전자(allele)의 빈도가 5% 이하인 SNPs는 제외시켰다. 다양한 SNPs를 포함하는 Enah 유전자 중 선택한 SNPs는 다음과 같다. 즉 rs487591, rs576861, rs755139, rs12034829, rs10799319, rs1891000, rs10915842, rs6681254, rs6751, rs2039620, rs1771316, rs1764345, rs954534, rs4653643, rs3795443 이다. Enah 유전자구역에 15개의 SNPs의 위치를 Fig. 1에 표시하였다. SNP 유전자형의 결정은 제공된 설명서에 따라 Illumina BeadStation 500 GX (Illumina Inc., San Diego, CA, USA)의 Golden Gate assay를 이용하여 시행하였다. 각각의 구형(bead type) 올리고 핵산염이 특이 SNP 좌위(locus)에 반응하였다. 각각의 Golden Gate 유전자형 결정은 99%를 넘는 정확도 재현비율(accuracy call rates)의 30 반복염기(30 replicates) 평균 강도에 해당되었다. 대립유전자의 발현빈도를 평가하고 연관성을 분석하였다.
3. 통계처리Enah 유전자 다형성과 가와사키병의 유전적 연관성을 분석하였다. 가와사키병군과 대조군 사이의 대립유전자 발현빈도를 SNPStats (http://bioinfo.iconcologia.net/index.php) [6]을 이용하여 비교하였다. 또한 가와사키병군 중에서 관상동맥 병변이 있는 군과 없는 군의 대립유전자 발현빈도를 비교하였다. 신뢰구간(confidence interval, CI) 95%의 교차비(odds ratio, OR), 해당 P-values를 얻고, 공통 변수로 성별에 의한 오차를 줄이기 위해 다발 로지스틱 회귀모델(multiple logistic regression models) 분석을 시행하였다. 유의수준은 P값 0.05 미만으로 하였다.
SNPStats을 이용하여 각 대립유전자가 하디 와인버그 평형(Hardy-Weinberg equilibrium)을 만족하는지 평가하였다.
각 대립유전자의 연관불균형(linkage disequilibrium)에 대한 확인은 Haploview software ver. 4.1 (Broad Institute, Cambridge, MA, USA)[7]을 이용하였다. 대조군과 가와사키병군 사이에 모든 SNPs 쌍에 대해 연관불균형 자료를 계산하였고 Enah 유전자의 다형성 사이에 구간(block)의 연관불균형과 일배체형(haplotype)을 분석하였다.
결 과가와사키병 환아와 건강 대조군의 임상적 특징은 Table 1에 표시된 바와 같았다. 가와사키병 환아의 남녀 비는 2.2:1, 평균 연령은 2년 7개월±2세였다. 106명 중 관상동맥 병변은 31명의 환아에서 관찰되었고, 75명은 없었다(Table 1).
SNPs 15개 중 1개(rs1891000)에서 가와사키병 발병과 의미 있는 연관성을 보였다. rs1891000(T/C)에서 TT, TC와 CC 유전형의 발현 빈도는 대조군에서 각각 66.7% (n=106), 32.7% (n=52), 0.6% (n=1)였고 가와사키병군에서는 33.3% (n=8), 66.7% (n=16), 0% (n= 0)였다. TT, TC와 CC 유전자형의 발현빈도는 가와사키병군이 대조군에 비해 3.25배 높았다. 이는 rs1891000의 소수 대립유전자(minor allele) C가 가와사키병의 발병에 위험인자로 작용한다는 것을 의미한다(codominant model, OR, 3.25; 95% CI, 1.36 to 7.79; P = 0.007; dominant model, OR, 3.59; 95% CI, 1.43 to 9.03; P= 0.005; overdominant model, OR, 3.67; 95% CI, 1.45 to 9.24; P= 0.005) (Table 2).
또한 관상동맥 병변의 발생과 Enah 유전자 다형성을 평가한 결과 15개의 SNPs 중 4개(rs487591, rs576861, rs7555139, rs10799319)에서 관상동맥 병변과 의미 있는 연관성을 보였다(rs487591: recessive model, P= 0.036; overdominant model, OR, 2.59; 95% CI, 1.07 to 6.26; P= 0.034; rs576861: recessive model, P= 0.036; rs7555139: recessive model, P= 0.036; rs10799319: recessive model, P= 0.036; overdominant model, OR, 2.49; 95% CI, 1.03 to 6.00; P= 0.040) (Table 3).
가브리엘 방법(Gabriel method)으로 Enah 유전자 15개 SNPs에서 한 개의 연관불균형 구간이 관찰되었다(Fig. 2). 유전자 SNPs을 쌍으로 비교해보던 중 각각에서 강한 연관불균형이 관찰되었다. 일배체형은 구간 1 (block 1)에 그려져 있다(Fig. 2). 구간의 일배체형은 다음과 같다. 즉 rs487591, rs576861, rs755139, rs12034829, rs10799319, rs6751, rs2039620, rs1771316, rs1764345, rs954534, rs4653643, rs3795443이다. 대조군과 가와사키병군의 일배체형 분석에서 연관불균형 구간 1에 빈도 0.1을 초과하는 일배체형은 Haploview software ver. 4.1 (Broad Institute)을 이용하여 분석하였고 두 군 간에 유의한 차이는 없었다(Table 4, Fig. 3).
고 찰가와사키병은 1967년 Kawasaki [8]가 처음으로 발표한 후 전 세계적으로 보고되어 왔다. 5세 이하의 어린 소아 연령에서 주로 발생하는 전신을 침범하는 급성 열성 혈관염으로 현재 소아에게서 발생하는 후천성 심질환의 가장 흔한 원인질환으로 알려져 있는데, 이는 주로 침범하는 혈관이 관상동맥으로 혈관염의 합병증으로 관상동맥의 확장 등 관상동맥 병변이 발생할 수 있기 때문이다. 치료하지 않으면 약 25% 정도에서 관상동맥류나 관상동맥의 확장이 발생하여 이로 인해 관상동맥의 혈전 및 협착이 생겨 협심증, 심근 경색 또는 급사의 원인이 되기도 한다[9].
전 세계적인 발생 분포를 보이지만 한국과 일본 같은 동아시아 지역에서 더 많이 발생한다. 국내에는 5세 이하 소아에서 연간 10만 명당 134.4명이 발병하여 일본의 239.6명에 이어 전 세계적으로 두 번째로 높은 발생 빈도를 보인다[10].
관상동맥염을 일으키는 데는 내피세포의 활성화, CD68 단핵구/대식세포, CD8 림프구, 희소클론 immunoglobulin A (IgA) 형질세포(oligoclonal IgA plasma cells)가 관련된 것으로 보인다[11]. 기질 금속단백분해효소(matrix metalloproteinase) 등과 같은 효소가 동맥벽 통합을 손상시켜 관상동맥류를 일으키는 데 관여한다[12]. 또한 혈관내피성장인자(vascular endothelial growth factor), 단핵구화학주성인자, 단핵구활성인자(monocyte chemotactic and activating factor), 종양괴사인자(tumor necrosis factor), 인터루킨 (interleukin) 등이 관계한다[13,14]. 활성 염증은 수주에서 수개월에 걸쳐 반흔 형성을 동반한 점진적 섬유화로 대치된다. 동맥의 재형성(remodeling) 과정에 혈관내피 증식이나 신생혈관 생성(neoangiogenesis)으로 협착이 생길 수 있다[15]. 관상동맥류는 시간이 경과함에 따라 변하여 심혈관조영술로 검사하였을 때 병을 앓고 1-2년 경과 후 약 50-67%에서 호전된다고 한다[16].
감염 등의 외부요인으로 촉발되어 면역매개혈관염(immune mediated vasculitis)인 가와사키병이 발병하는 데는 유전적 소인이 중요할 것으로 보인다. 특정 연령층에 이환되고, 지역적 및 계절적 호발현상이 있어 어떤 감염과 관련이 있을 것으로 추측되지만 특정한 병원체는 발견되지 않았다. 아시아인에 발병률이 높으며, 같은 연령대의 소아와 비교해서 형제 간 발생 위험도가 높은 것으로 보아 유전적 요인이 있는 것으로 추측된다[17].
최근 가와사키병의 유전적 감수성을 연구하는데 인간 유전체 사업(human genome project)이 유용한 도구로 이용되고 있으며, 가와사키병의 발병이나 관상동맥류 발생에 취약한 유전자 다형성에 대한 연구가 점차 늘어나고 있다[3].
가와사키병은 하나의 유전자 이상으로 생기는 단일유전자질환과는 달리 감염 등 환경적 소인과 유전적 소인이 복합적으로 작용하는 복합성 질환에 속한다. 후보 유전자 연관 연구는 질환군과 대조군에서 후보유전자 다형성의 대립유전자 빈도의 차이를 밝혀서 대립유전자와 질병과의 연관관계를 찾는 방법으로[18], 본 연구는 Enah 유전자를 후보유전자로 하여 연관 연구를 시행하였다.
유전자가 존재하는 염색체상의 부위를 유전자 좌위라고 한다. 유전자에 변이가 있을 경우 같은 유전자 좌위에 복수의 유전자가 존재하게 되는데 이를 대립유전자라고 한다. 30억 염기(nucleotide) 쌍으로 이루어진 인간 게놈 유전자 염기서열은 99.9% 동일하며 개체 간에 0.1%의 차이가 있는 것으로 알려져 있다. 다형성(polymorphism)이란 개인 사이의 DNA에 존재하는 염기의 차이를 말하는데, 대립유전자의 빈도가 1% 이상에서 관찰될 때 다형성이라고 정의한다. 이 중 SNP가 가장 흔하고 약 1,000만 개의 SNPs가 존재하는 것으로 추정된다[19].
Enah 유전자는 1992년에 Sazuka 등[20]이 쥐 배아의 뇌 발달 동안 억제되어 있는 mRNA의 cDNA를 분리하면서 처음으로 발견되었다. 이를 NDPP-1으로 명명하였고 쥐의 중추신경계 발달에 중요한 역할을 하는 유전자로 추측하였다. Ena, mammalian Ena (Mena), drosophila로 불리기도 하며 사람의 1번 염색체 장완(long arm, 1q42.12)에 위치하고[21], 크기는 157 kb이고 14 엑손(exon)으로 이루어져있다[22].
Enah 유전자가 암호화하는 단백은 Ena/VASP이다. Ena/VASP 는 액틴 세포뼈대의 주요 조절자로 차차 여러 질환에 연관성이 밝혀지고 있어 생체의학연구의 유망한 표적으로 부상되고 있다[4]. 척추동물에서 Ena/VASP 군은 세 개의 구성원 즉, Mena, VASP, Ena VASP-like로 이루어져 있다. 이 중 VASP는 인체에서 혈소판을 프로스타글란딘(prostaglandin)이나 일산화질소(nitric oxide) 공여자와 같은 혈관확장제로 자극하여 최초로 분리된 인산화단백으로 다양한 세포에 존재하지만 특히 혈소판, 평활근근육세포, 혈관내피세포 등에 현저하게 발현된다고 알려져있다[23].
Ena/VASP의 구조는 N-말단(terminal) Ena/VASP 상동(homology) 1 (EVH1) 영역, 중앙부 프롤린 풍부 영역(central proline rich domain), C-말단 Ena/VASP 상동 2 (EVH2) 영역으로 이루어지는 공통의 세 영역(domain)을 가지며 기능 면에서도 서로 겹치는 부분이 있다[24]. EVH1 영역 결합부위는 다양한 단백에 존재하며 Ena/VASP가 수용체에 동원되는 것을 매개한다. EVH2 영역은 액틴에 결합하는 부위로, 모자 단백(capping protein)으로 보호되어 있는 액틴의 모자를 제거하여 Ena/VASP가 액틴(actin) 잔섬유를 연장하는 역할을 하는 부분이다. Ena/VASP는 주로 c-AMP-dependent protein kinase와 c-GMP-dependent protein kinase에 의해 인산화되면서 활성화되어 다양한 조직에서 세포의 이동과 부착, 성장의 조절에 관여한다[25].
세포뼈대는 세포질에 들어있는 세포골격으로 세포의 모양을 유지하고, 보호하며 이동을 가능하게 한다. 또한 세포 내의 물질 수송과 세포분열에 중요한 역할을 한다. 진핵세포생물의 세포질에는 미세섬유(microfilament), 중간 잔섬유(intermediate filament), 미세관(microtubule)이라는 세 가지 종류의 주요 세포뼈대가 들어 있다. 이 중 미세섬유는 두 줄의 액틴 사슬이 꼬여서 만들어지며 세포 막 바로 밑에 존재하여 장력에 저항하여 세포 모양을 유지하고 세포돌기(protuberance)를 만들어 세포와 세포 혹은 세포와 기질 결합에 관여한다. 결합에 관여하는 역할로 미세섬유는 전달에 필수적이며 또한 세포질 분열에 중요하고 미오신(myosin)과 함께 근육 수축에 관여한다[26].
미세섬유를 만드는 액틴 세포뼈대는 면역반응, 혈관의 유지, 지혈 등과 같은 인체 항상성 유지에 관계한다. 따라서 액틴 세포뼈대의 기능은 엄격하게 조절되어야 하며 비정상으로 조절되면 질병이 발생할 수 있다. 액틴 세포뼈대에 결합하여 이를 조절하는 많은 단백 중 하나가 Ena/VASP로 항모자형성(anti-capping)의 기능을 가져서 액틴 미세구조를 직접 조정하여 세포 모양의 변화, 부착, 이동, 세포 간 상호작용 등 중요한 기능에 관여한다. Ena/VASP와 연관된 질병으로는 동맥경화, 심근증, 암, 혈전증, 사구체신염 등이 보고되어있다[4].
Ena/VASP는 모든 세포 내에 존재하며 세포의 활성화, 부착, 투과성, 이동, 혈관생성에 관련되는 액틴 잔섬유의 변화(dynamics)를 조절하는 단백이다. 특히 혈소판, 혈관내피세포에 현저하게 발현되어 혈소판과 혈관내피세포의 세포뼈대 조절자 역할로 혈관염의 발생과 깊은 관련을 갖고 있을 것으로 보여 전신적 혈관염인 가와사키병의 발병 및 혈관염의 합병증인 관상동맥 병변의 발생에 기여할 가능성이 있다고 추측된다.
저자의 연구는 Enah 유전자와 가와사키병과 그 합병증인 관상 동맥류 발생의 연관성을 알아보기 위한 최초의 연구로 rs1891000 (T/C)에서 소수 대립유전자(minor allele) C는 가와사키병의 발병에 위험인자로 작용함을 관찰하였다. 또한 관상동맥 병변의 발생과 Enah 유전자 다형성의 연관성을 평가한 결과 15개의 SNPs 중 4개(rs487591, rs576861, rs7555139, rs10799319)에서 관상동맥 병변과 의미 있는 연관성을 보였다. 아울러 의미가 있게 나온 SNPs rs487591, rs576861, rs7555139, rs10799319에서 소수 대립유전자의 동형접합자(homozygote)는 관상동맥 병변이 있는 군에서는 발현되지 않았고 관상동맥 병변이 없는 군에서 모두 8%의 빈도로 발현되어 소수 대립유전자의 동형접합자가 관상동맥 병변이 덜 생기게 보호할 가능성을 조심스럽게 추측해 볼 수 있었다.
본 연구는 표본 수가 적은 자료로 성급하게 결론을 내릴 수는 없고 차후에 더 많은 표본으로 연구를 하여 확인해볼 필요가 있다고 생각된다. 또한 최초의 연구이므로 기존의 연구에 의해 알려진 정보가 많지 않아 Enah/VASP의 가와사키병과 관상동맥 병변을 유발시키는 경로를 확연하게 규명하기에는 무리가 있으며 Enah/ VASP 등의 발현을 동시에 검사하는 등의 임상적 연결에 대한 연구가 같이 시행되지 못한 제한점을 갖는다. 차후 이러한 문제점을 다기관, 인종의 연구 및 다른 방법의 Enah/VASP의 연구를 통해 보완해나가야 할 것으로 보인다.
Fig. 1.Locations of selected single nucleotide polymorphisms in the enabled homolog gene on human chromosome 1. 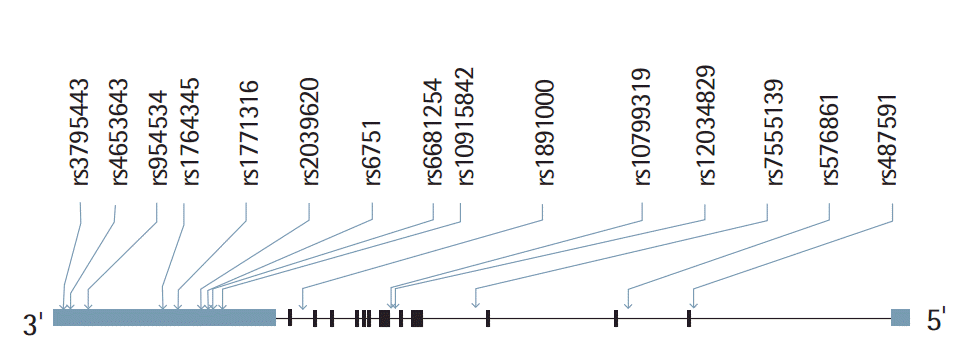
Fig. 2.Block of linkage disequilibrium in the 15 selected single nucleotide polymorphisms of enabled homolog. 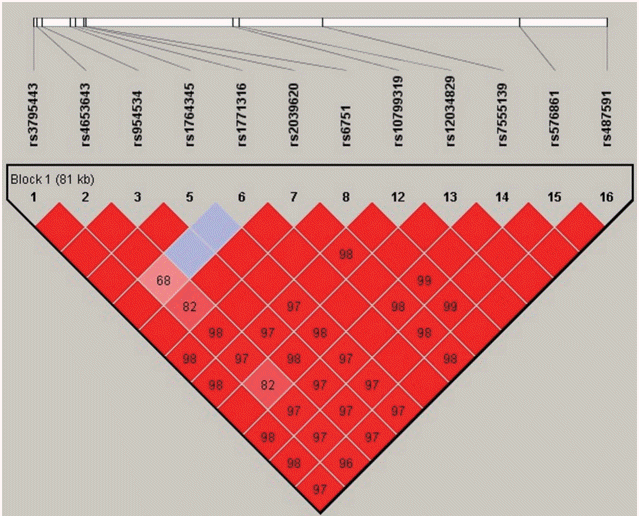
Fig. 3.Haplotype frequency of linkage disequilibrium in the 15 selected single nucleotide polymorphisms of enabled homolog. 
Table 1.Demographics of the patients with Kawasaki disease with or without coronary artery lesions and control subjects
Table 2.Logistic analysis of the selected 15 single nucleotide polymorphisms of enabled homolog in patients with Kawasaki disease and healthy control subjects
Table 3.Logistic analysis of the selected 15 single nucleotide polymorphisms of enabled homolog in Kawasaki disease with or without coronary artery disease
Table 4.Haplotype association of blocked single nucleotide polymorphisms in enabled homolog REFERENCES1. Newburger JW, Takahashi M, Gerber MA, Gewitz MH, Tani LY, Burns JC, et al. Diagnosis, treatment, and long-term management of Kawasaki disease: a statement for health professionals from the Committee on Rheumatic Fever, Endocarditis, and Kawasaki Disease, Council on Cardiovascular Disease in the Young, American Heart Association Pediatrics 2004;114: 1708-33.
2. Wang CL, Wu YT, Liu CA, Kuo HC, Yang KD. Kawasaki disease: infection, immunity and genetics. Pediatr Infect Dis J 2005;24: 998-1004.
3. Lee JK, Hong YM, Jang GY, Yun SW, Yu JJ, Yoon KL, et al. ConsortiumBased Genetic Studies of Kawasaki Disease in Korea: Korean Kawasaki Disease Genetics Consortium. Korean Circ J 2015;45: 443-8.
4. Pula G, Krause M. Role of Ena/VASP proteins in homeostasis and disease. Handb Exp Pharmacol 2008;(186):39-65.
6. Sole X, Guino E, Valls J, Iniesta R, Moreno V. SNPStats: a web tool for the analysis of association studies. Bioinformatics 2006;22: 1928-9.
7. Barrett JC, Fry B, Maller J, Daly MJ. Haploview: analysis and visualization of LD and haplotype maps. Bioinformatics 2005;21: 263-5.
8. Kawasaki T. Acute febrile mucocutaneous syndrome with lymphoid involvement with specific desquamation of the fingers and toes in children. Arerugi 1967;16: 178-222.
10. Kim GB, Han JW, Park YW, Song MS, Hong YM, Cha SH, et al. Epidemiologic features of Kawasaki disease in South Korea: data from nationwide survey, 2009-2011. Pediatr Infect Dis J 2014;33: 24-7.
11. Rowley AH, Shulman ST, Spike BT, Mask CA, Baker SC. Oligoclonal IgA response in the vascular wall in acute Kawasaki disease. J Immunol 2001;166: 1334-43.
12. Takeshita S, Tokutomi T, Kawase H, Nakatani K, Tsujimoto H, Kawamura Y, et al. Elevated serum levels of matrix metalloproteinase-9 (MMP-9) in Kawasaki disease. Clin Exp Immunol 2001;125: 340-4.
13. Yasukawa K, Terai M, Shulman ST, Toyozaki T, Yajima S, Kohno Y, et al. Systemic production of vascular endothelial growth factor and fms-like tyrosine kinase-1 receptor in acute Kawasaki disease. Circulation 2002;105: 766-9.
14. Lin CY, Lin CC, Hwang B, Chiang B. Serial changes of serum interleukin-6, interleukin-8, and tumor necrosis factor alpha among patients with Kawasaki disease. J Pediatr 1992;121: 924-6.
15. Suzuki A, Miyagawa-Tomita S, Komatsu K, Nishikawa T, Sakomura Y, et al. Active remodeling of the coronary arterial lesions in the late phase of Kawasaki disease: immunohistochemical study. Circulation 2000;101: 2935-41.
16. Kato H, Sugimura T, Akagi T, Sato N, Hashino K, Maeno Y, et al. Longterm consequences of Kawasaki disease: a 10- to 21-year follow-up study of 594 patients. Circulation 1996;94: 1379-85.
18. Newton-Cheh C, Hirschhorn JN. Genetic association studies of complex traits: design and analysis issues. Mutat Res 2005;573: 54-69.
20. Sazuka T, Tomooka Y, Kathju S, Ikawa Y, Noda M, Kumar S. Identification of a developmentally regulated gene in the mouse central nervous system which encodes a novel proline rich protein. Biochim Biophys Acta 1992;1132: 240-8.
21. Gregory SG, Barlow KF, McLay KE, Kaul R, Swarbreck D, Dunham A, et al. The DNA sequence and biological annotation of human chromosome 1. Nature 2006;441: 315-21.
22. Urbanelli L, Massini C, Emiliani C, Orlacchio A, Bernardi G, Orlacchio A. Characterization of human Enah gene. Biochim Biophys Acta 2006;1759: 99-107.
23. Halbrügge M, Walter U. Purification of a vasodilator-regulated phosphoprotein from human platelets. Eur J Biochem 1989;185: 41-50.
24. Gertler FB, Niebuhr K, Reinhard M, Wehland J, Soriano P. Mena, a relative of VASP and Drosophila Enabled, is implicated in the control of microfilament dynamics. Cell 1996;87: 227-39.
|
|
|||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||